La notizia è di settimana scorsa, ma non ne avevo ancora parlato sul blog perché quando si tratta di scoperte così bizzarre preferisco documentarmi un attimo e leggere l’articolo originale, pubblicato in questo caso sulla prestigiosa rivista PNAS. Secondo uno studio condotto da ricercatori americani, infatti, sembra esistere una correlazione tra i nostri geni e gli amici che ci scegliamo!
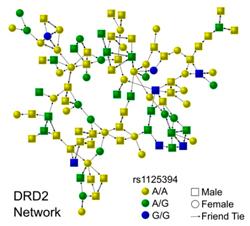
Gli autori hanno analizzato sei geni legati al comportamento (DRD2, DRD4, CYP2A6, MAOA, SLC6A3 e SLC6A4) in una rete sociale composta da circa un migliaio di individui, scoprendo che due di essi (DRD2 e CYP2A6) mostravano una correlazione piuttosto evidente con la scelta delle amicizie. In particolare, due persone amiche tra loro avevano un’alta probabilità di condividere la stessa variante genetica del gene DRD2, mentre per il CYP2A6 la correlazione era inversa: gli individui cercavano amici con una variante del gene diversa dalla propria. Le correlazioni sono state confermate anche in una seconda rete di amicizie composta da 2000-3000 soggetti.

Leggendo l’articolo si scopre che le associazioni trovate sembrano piuttosto forti e statisticamente significative. Per quanto riguarda DRD2 il p-value è di 0.0001: vuol dire che c’è appena una probabilità su 10mila che la correlazione trovata sia dovuta al caso. Esiste quindi davvero una stretta associazione tra questi due geni e il modo in cui selezioniamo i nostri amici, anche se dare un’interpretazione convincente a questo risultato non è affatto semplice. Gli autori ritengono che il caso del gene DRD2 possa essere spiegato in funzione dell’associazione di questo gene con l’alcolismo: chi ha una variante che lo predispone all’alcolismo cercherà amici con la stessa variante, con cui ubriacarsi in compagnia. Un po’ meno intuitivo è il discorso relativo al gene CYP2A6: chi possiede una certa variante di questo gene è caratterialmente più aperto, quindi non si riesce a capire perché una persona “geneticamente” più socievole debba volontariamente escludere persone altrettanto espansive dalla propria cerchia di amici.
Come sempre accade quando si parla di genetica comportamentale, non sono mancate critiche e commenti scettici. Il genetista David Altshuler, del Broad Institute di Cambridge, ricorda che gli studi di associazione genome-wide che vengono normalmente condotti per trovare le basi genetiche delle malattie devono analizzare centinaia di migliaia di varianti per riuscire a individuarne qualcuna significativa. Trovare due geni buoni analizzandone solo sei – dice Altshuler – è come comprare sei biglietti della lotteria e vincere il primo e il secondo premio: gli autori sono stati troppo fortunati, così fortunati da far pensare che ci sia sotto qualcosa. Eppure le due associazioni trovate sono statisticamente robuste: i p-value sono significativi. Quindi? Dove sta l’inghippo?
Mentre leggevo l’articolo con lo stesso sano scetticismo di Altshuler, mi è caduto l’occhio su tre paroline magiche: “correzione di Bonferroni“. Questa procedura si utilizza per eliminare i falsi positivi quando si compiono una serie di test statistici, e dipende direttamente dal numero di test eseguiti. Non voglio entrare nei dettagli (chi conosce un po’ di statistica avrà capito di cosa sto parlando), ma in pratica più test eseguo (cioè più geni vado a testare) più devo essere stringente nella mia soglia di significatività. Se fisso una soglia iniziale di significatività a 0.05, la soglia corretta con il metodo di Bonferroni sarà 0.05 diviso 6 (0.008): tutti i geni che hanno un p-value inferiore a 0.008 saranno considerati buoni. Se però invece di fare 6 test ne faccio 100mila, l’asticella si abbasserà di tantissimo: il p-value massimo accettato sarà 5 per dieci alla meno 7, molto più basso dei valori trovati in questo studio!
Questa digressione statistica soltanto per dimostrarvi che se avessero eseguito un GWAS come Dio comanda, con centinaia di migliaia di SNP da testare, molto probabilmente DRD2 e CYP2A6 non li avrebbero mai trovati. James Fowler, uno degli autori, ha dichiarato sul blog Genetic Future che presto avranno i risultati di un vero studio di associazione genome-wide basato su 17mila individui: sarà un ottimo banco di prova per le due associazioni trovate. Per il momento io rimango col beneficio del dubbio.
Fowler, J., Settle, J., & Christakis, N. (2011). Correlated genotypes in friendship networks Proceedings of the National Academy of Sciences DOI: 10.1073/pnas.1011687108