Bisogna ammetterlo, gli organizzatori del Carnevale della Biodiversità conoscono il gusto della provocazione. Dopo “Le dimensioni contano”, “Nicchie estreme: ai confini della realtà” e “Alieni tra noi”, questa volta i partecipanti hanno affrontato un tema ancor più originale, che li ha costretti a spremere le meningi e a mettere sul campo tutta la loro creatività e fantasia. Il titolo di questa edizione del carnevale, tornato dopo una lunga pausa, ha infatti una connotazione fantascientifica piuttosto che scientifica: “Ho visto cose.. La biologia dei mondi fantastici”. Non appena l’ho letto, nella mia testa è scattato il collegamento con la biologia sintetica, un settore che ho iniziato a conoscere da vicino qualche mese fa, durante la mia visita all’ETH di Basilea. Che cosa c’è infatti di più fantastico di una biologia fabbricata dall’uomo, che in natura non esiste? Dopotutto, fare biologia sintetica significa realizzare molecole e organismi che non sono mai esistiti, e che tuttavia sarebbero potuti esistere se l’evoluzione avesse preso un altro corso. Fare biologia sintetica è un po’ come sbirciare in un mondo immaginario, un mondo che per la maggioranza di noi sarà popolato da draghi, fate e supereroi, ma che per un biologo molecolare potrebbe ospitare forme di vita ancora più bizzarre. Organismi diversi da quelli che conosciamo non solo per il loro aspetto o per il loro genoma, ma addirittura per il codice genetico in base al quale sono stati programmati.
Bisogna ammetterlo, gli organizzatori del Carnevale della Biodiversità conoscono il gusto della provocazione. Dopo “Le dimensioni contano”, “Nicchie estreme: ai confini della realtà” e “Alieni tra noi”, questa volta i partecipanti hanno affrontato un tema ancor più originale, che li ha costretti a spremere le meningi e a mettere sul campo tutta la loro creatività e fantasia. Il titolo di questa edizione del carnevale, tornato dopo una lunga pausa, ha infatti una connotazione fantascientifica piuttosto che scientifica: “Ho visto cose.. La biologia dei mondi fantastici”. Non appena l’ho letto, nella mia testa è scattato il collegamento con la biologia sintetica, un settore che ho iniziato a conoscere da vicino qualche mese fa, durante la mia visita all’ETH di Basilea. Che cosa c’è infatti di più fantastico di una biologia fabbricata dall’uomo, che in natura non esiste? Dopotutto, fare biologia sintetica significa realizzare molecole e organismi che non sono mai esistiti, e che tuttavia sarebbero potuti esistere se l’evoluzione avesse preso un altro corso. Fare biologia sintetica è un po’ come sbirciare in un mondo immaginario, un mondo che per la maggioranza di noi sarà popolato da draghi, fate e supereroi, ma che per un biologo molecolare potrebbe ospitare forme di vita ancora più bizzarre. Organismi diversi da quelli che conosciamo non solo per il loro aspetto o per il loro genoma, ma addirittura per il codice genetico in base al quale sono stati programmati.
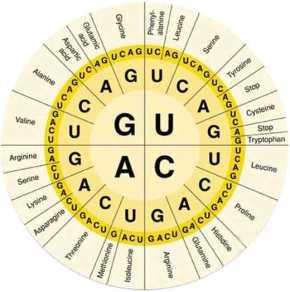
Fabbricare una proteina è un processo sofisticato nel quale intervengono diversi attori. Il ruolo del protagonista appartiene indubbiamente al ribosoma, un complesso macchinario composto da tre molecole di RNA e da più di 50 proteine. Spetta al ribosoma effettuare la sintesi vera e propria, andando a decodificare la sequenza nucleotidica dell’RNA messaggero in arrivo dal nucleo. I codoni vengono fatti scorrere, l’uno dopo l’altro, pronti per essere letti. Ed è qui che interviene l’altra molecola decisiva in questo processo, la chiave di lettura che consente al ribosoma di interpretare l’informazione portata dal messaggero e trasformarla in una catena di aminoacidi: sono i tRNA (o RNA transfer). Questi RNA di trasporto raggiungono il ribosoma e vanno ad appaiarsi con il codone su cui questo sta transitando. Se il codone si lega di buon grado all’anticodone portato dal tRNA, ecco che avviene la magia e il tRNA cede il suo prezioso carico: un aminoacido, che viene immediatamente trasferito alla catena proteica nascente. Il processo va avanti finché il ribosoma non incontra uno dei tre codoni di stop (UAA, UAG, UGA), che non riescono ad appaiarsi ad alcun tRNA. La traduzione funziona perché ogni codone può appaiarsi a un solo tRNA, e quel tRNA porterà sempre con sé lo stesso identico aminoacido. Già, ma una volta compiuto il loro dovere, che fanno i tRNA? Bussano alla porta degli enzimi amminoacil-tRNA sintetasi, che li ricaricano consegnando loro un nuovo aminoacido. Esistono 20 tipi diversi di questo enzima, ognuno competente per uno specifico aminoacido. Per cambiare il codice genetico e scriverne uno nuovo occorre dunque intervenire su tutti e tre questi componenti: il ribosoma, i tRNA e l’aminoacil-tRNA sintetasi. E’ quello che stanno cercando di fare all’Università di Cambridge, nel gruppo di ricerca guidato da Jason W. Chin. Il primo passo è stato creare un ribosoma alternativo a quello naturale, vediamo come hanno fatto.


A onor del vero, in passato erano già stati condotti esperimenti in cui si cercava di introdurre aminoacidi non naturali sfruttando le quadriplette al posto delle triplette, ma con i ribosomi naturali questa operazione risultava complicata: i tRNA speciali in grado di appaiarsi a codoni di quattro lettere fanno fatica a entrare nel ribosoma, e d’altra parte ogni tentativo di aumentare l’efficienza di traduzione rischiava di danneggiare in modo letale tutte le altre proteine. Con il ribosoma alternativo messo a punto dal gruppo di Jason W. Chin non c’era più questo pericolo. I ricercatori hanno quindi testato oltre un miliardo di ribosomi leggermente diversi tra loro, con l’obiettivo di trovarne uno che fornisse un alloggio più comodo per i tRNA speciali. Ancora una volta hanno costretto le cellule a fabbricare la proteina che dava resistenza al cloramfenicolo, ma in questo caso il gene che la codificava aveva in un certo punto un codone di quattro lettere, che poteva essere letto solo se il ribosoma riusciva ad ospitare un apposito tRNA speciale introdotto dai ricercatori. E’ bastato aggiungere un po’ di antibiotico per fare fuori tutte le cellule prive di questo superpotere e scovare il ribosoma in grado di leggere i codoni da quattro lettere tanto quanto quelli da 3: lo hanno chiamato Ribo-Q1.
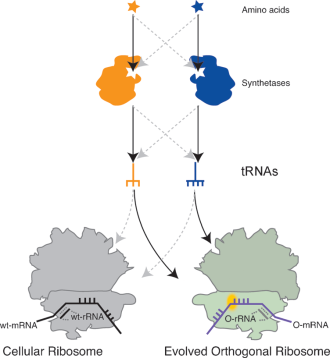
Missione compiuta dunque? Non proprio. In natura esistono solo due coppie sintetasi/tRNA realmente ortogonali al sistema di traduzione di E. coli: sono le uniche che non disturbano la normale attività di traduzione, e la sola cosa che posso fare è cambiare i due aminoacidi associati a queste due coppie. Questo significa che è impossibile inserire in una proteina più di due aminoacidi non naturali! Ecco dunque la vera sfida per le prossime ricerche: se vogliamo produrre polimeri completamente non naturali, bisogna escogitare strategie per sviluppare nuove coppie sintetasi/tRNA disponibili ad accettare altri aminoacidi. Se i biologi sintetici riusciranno nell’impresa, in futuro potremmo fare esperimenti molto interessanti e scoprire che, con qualche aminoacido in più a disposizione, la vita potrebbe evolvere funzioni nuove e imprevedibili, funzioni che per la biologia naturale sono fuori portata. Ma qui si entra nel regno della fantascienza, anzi, si entra nella biologia dei mondi fantastici. Trovate gli altri contributi al Carnevale della Biodiversità sul blog Mahengechromis. Buona lettura!
Wang, K., Schmied, W., & Chin, J. (2012). Reprogramming the Genetic Code: From Triplet to Quadruplet Codes Angewandte Chemie International Edition, 51 (10), 2288-2297 DOI: 10.1002/anie.201105016